A rinossinusite crônica é uma doença multifatorial cuja patogênese, influenciada por fatores genéticos e ambientais, ainda é incerta. Estudos genéticos anteriores mostraram que pacientes com rinossinusite crônica têm expressão reduzida do gene da Interleucina‐22 (IL‐22).
ObjetivoIdentificar e comparar a frequência de polimorfismos no gene IL22RA1 (receptor subunidade alfa 1da interleucina 22) entre os pacientes com rinossinusite crônica com ou sem pólipos nasais.
MétodoAmostras de sangue periférico foram coletadas de 70 pacientes com rinossinusite crônica com pólipos nasais, 14 pacientes com rinossinusite crônica sem pólipos nasais e 68 indivíduos sem rinossinusite crônica, seguido por extração de DNA e análise da sequência do gene IL22RA1.
ResultadosDos dez polimorfismos identificados no gene IL22RA1, três não foram encontrados em alguma banco de dados genético analisado. Pacientes com rinossinusite crônica apresentaram maior frequência da inserção frameshift c.113_114insA, possivelmente patogênica. Por outro lado, no grupo controle, o polimorfismo c.435A>C mostrou predominância significativa do alelo mutado, talvez relacionado a uma proteção potencial contra o fenótipo da rinossinusite crônica. O polimorfismo c.770C> T, caracterizado como uma variante não sinônima, foi encontrado exclusivamente em pacientes negros com rinossinusite crônica com pólipos nasais.
ConclusõesEmbora nenhuma relação causal direta possa ser estabelecida entre os polimorfismos do gene IL22RA1 e a fisiopatologia da rinossinusite crônica, variações genéticas como c.113_114insA e c.435A>C podem estar envolvidas na suscetibilidade ou proteção contra o fenótipo da rinossinusite crônica, respectivamente. Testar essa hipótese exigirá estudos com coortes maiores.
Apesar da identificação de vários fatores potencialmente envolvidos na rinossinusite crônica (RSC) – inclusive respostas alérgicas, comprometimento de clearance mucociliar, disfunção imune, alteração da defesa epitelial, agentes microbianos e exposição ambiental1 – sua etiologia permanece incerta. Um manejo mais assertivo dos pacientes requer esclarecimentos adicionais sobre como esses fatores interagem entre si, bem como os fatores genéticos na etiologia da RSC.
A interleucina 22 (IL‐22) é uma citocina da família IL‐10 que pode estar envolvida na resposta inflamatória da fase aguda, ativação do sistema imune inato, migração celular induzida, inibição das células dendríticas e atenuação das respostas alérgicas.1,2 O receptor IL22RA1 também pode desempenhar um papel essencial na cicatrização e reorganização dos tecidos. Kotenko et al.3 mostraram que os níveis de IL22RA1 eram significativamente mais baixos nas células epiteliais de pacientes com RSC com pólipos nasais (RSCcPN) que não responderam à cirurgia. Isso pode resultar em danos à barreira epitelial, com uma diminuição na produção de citocinas pró‐inflamatórias Th1, causar uma inversão no padrão de resposta Th1/Th2. Alternativamente, alterações no regulador do complexo heterodímero (IL22RA1‐IL10R2, IL20 e IL24) podem levar à diminuição da resposta imune inata (β‐defensinas, mucinas e metaloproteinases), aumento dos níveis de citocinas pró‐inflamatórias (IL‐6, IL‐8 e TNF) e redução da apoptose,3,4 fornecer outros possíveis mecanismos fisiopatológicos envolvidos no desenvolvimento da RSC.
Embora as variantes genéticas possam não ser a origem de uma determinada doença, elas podem conferir suscetibilidade ao seu desenvolvimento. Portanto, o presente estudo teve como objetivo identificar variantes associadas ao gene IL22RA1 em pacientes brasileiros com diagnóstico de rinossinusite crônica e em indivíduos sem esse diagnóstico.
MétodoA escolha do gene IL22RA1 resultou de uma pesquisa canadense anterior sobre agrupamento de DNA (Genome‐Wide Association Study)5 que selecionou cinco polimorfismos de nucleotídeo único (SNPs) de alta prioridade e outros 18 polimorfismos do projeto internacional HapMap.6 Entre esses 23 SNPs, o gene IL22RA1 foi um forte candidato ao desenvolvimento da RSC.5
O presente estudo foi aprovado pelo comitê de ética em pesquisa do Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo (HCFMRP‐USP) e pela Comissão Nacional de Ética em Pesquisa (Conep), sob protocolo n° 5243/2011. Todos os pacientes e controles foram convidados a participar da pesquisa após esclarecimento dos procedimentos e objetivos do estudo, método de coleta de amostra, riscos e possível acesso aos resultados, conforme indicado em um termo de consentimento livre e esclarecido (TCLE).
Pacientes e controles foram recrutados no ambulatório de otorrinolaringologia do HCFMRP‐USP. De acordo com os critérios clínicos da diretriz do European Position Paper on Rhinosinusitis and Nasal Polyps (EPOS) de 2012,7 os pacientes foram classificados como portadores de RSC com ou sem PN. O grupo controle foi constituído por 68 voluntários sem sintomas nasais e com nasofibroscopia normal, não relacionados aos participantes dos outros dois grupos. Todos os participantes preencheram um questionário com nome, idade, sexo, origem atual e remota, tabagismo, cor da pele e histórico atual de doenças, inclusive informações sobre alergias sazonais e perenes, asma e intolerância ao ácido acetilsalicílico. Pacientes com síndrome de Churg‐Strauss, discinesia ciliar primária e fibrose cística foram excluídos do estudo.
O plasma foi extraído das amostras de sangue periférico por centrifugação a 2500 RPM por 10 minutos a 4°C. Depois, o DNA foi extraído da camada leucocitária (Buffy Coat). Os sete éxons do gene IL22RA1 foram amplificados por reação em cadeia de polimerase (PCR), com o sétimo exon dividido em cinco sequências, devido ao seu tamanho, para simplificar a análise. O protocolo da PCR incluiu desnaturação a 95°C, seguida de annealing, a temperaturas escolhidas de acordo com o primer usado e extensão de DNA a 72° C, repetida por aproximadamente 35 ciclos até atingir o platô da reação (fase final). Os produtos da PCR foram analisados em gel de agarose a 1,5% para confirmar a amplificação e o tamanho esperado do fragmento.
Cada exon foi sequenciado no sequenciador automático 3500 XL Genetic Analyzer (Applied Biosystems) com o método Sanger. O arquivo de saída do sequenciador consiste em um cromatograma que contém as informações da sequência e a qualidade dos nucleotídeos.
A patogenicidade das variantes foi investigada com a ferramenta UMD‐Predictor,8 que combina propriedades bioquímicas de aminoácidos mutados, impacto nos sinais de splicing, localização de mutações em domínios funcionais, frequência de variantes na população global, conservação de aminoácidos mutados com a matriz de substituição global BLOSUM62 e conservação de sequências de aminoácidos em 100 espécies. Os preditores PolyPhen29 e Sift/PROVEAN10 foram usados para auxiliar os resultados da UMD‐Predictor. As variantes também foram analisadas com o algoritmo CADD (Combined Annotation Dependent Depletion), que avalia o grau de patogenicidade da mutação, inclusive pequenas inserções e deleções (InDels).11 A plataforma VarSome foi usada para consolidar todas as informações relacionadas à previsão de patogenicidade.12
O teste exato de Fisher foi usado para avaliar possíveis associações entre as variáveis analisadas. A análise dos polimorfismos foi feita em diferentes etapas: grupo RSCcPN versus grupo controle, grupo RSCsPN versus grupo controle e grupo RSCcPN versus grupo RSCsPN (tabelas 1‐3).
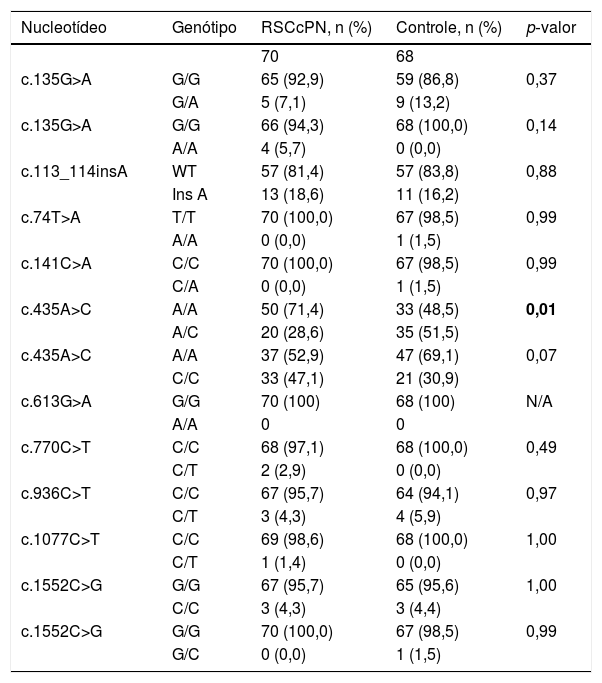
Análise estatística que compara as frequências alélicas em pacientes com rinossinusite crônica com pólipos nasais e indivíduos controle
| Nucleotídeo | Genótipo | RSCcPN, n (%) | Controle, n (%) | p‐valor |
|---|---|---|---|---|
| 70 | 68 | |||
| c.135G>A | G/G | 65 (92,9) | 59 (86,8) | 0,37 |
| G/A | 5 (7,1) | 9 (13,2) | ||
| c.135G>A | G/G | 66 (94,3) | 68 (100,0) | 0,14 |
| A/A | 4 (5,7) | 0 (0,0) | ||
| c.113_114insA | WT | 57 (81,4) | 57 (83,8) | 0,88 |
| Ins A | 13 (18,6) | 11 (16,2) | ||
| c.74T>A | T/T | 70 (100,0) | 67 (98,5) | 0,99 |
| A/A | 0 (0,0) | 1 (1,5) | ||
| c.141C>A | C/C | 70 (100,0) | 67 (98,5) | 0,99 |
| C/A | 0 (0,0) | 1 (1,5) | ||
| c.435A>C | A/A | 50 (71,4) | 33 (48,5) | 0,01 |
| A/C | 20 (28,6) | 35 (51,5) | ||
| c.435A>C | A/A | 37 (52,9) | 47 (69,1) | 0,07 |
| C/C | 33 (47,1) | 21 (30,9) | ||
| c.613G>A | G/G | 70 (100) | 68 (100) | N/A |
| A/A | 0 | 0 | ||
| c.770C>T | C/C | 68 (97,1) | 68 (100,0) | 0,49 |
| C/T | 2 (2,9) | 0 (0,0) | ||
| c.936C>T | C/C | 67 (95,7) | 64 (94,1) | 0,97 |
| C/T | 3 (4,3) | 4 (5,9) | ||
| c.1077C>T | C/C | 69 (98,6) | 68 (100,0) | 1,00 |
| C/T | 1 (1,4) | 0 (0,0) | ||
| c.1552C>G | G/G | 67 (95,7) | 65 (95,6) | 1,00 |
| C/C | 3 (4,3) | 3 (4,4) | ||
| c.1552C>G | G/G | 70 (100,0) | 67 (98,5) | 0,99 |
| G/C | 0 (0,0) | 1 (1,5) |
RSCcPN, rinossinusite crônica com pólipos nasais; n, número de amostras; C, citosina; T, timina; A, adenina; G, guanina; WT, tipo selvagem, N/A, não aplicável.
p < 0,01: possível proteção contra os fenotípicos.
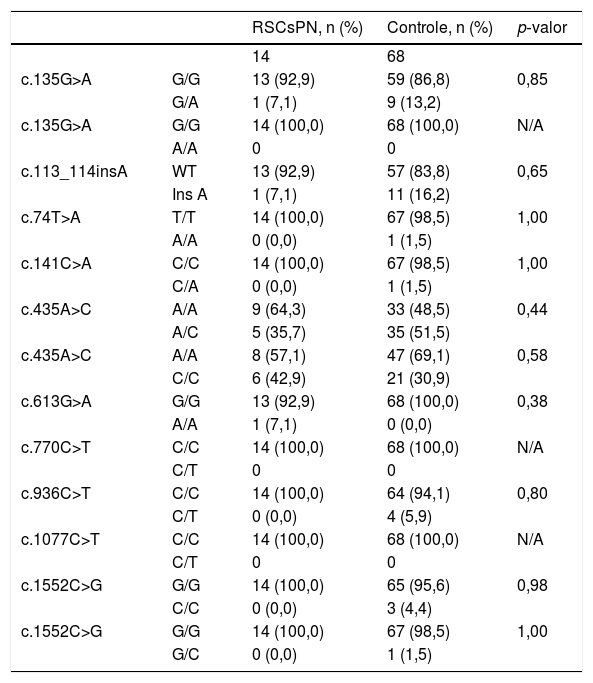
Análise estatística que comparando as frequências alélicas em pacientes com rinossinusite crônica sem pólipos nasais e indivíduos controle
| RSCsPN, n (%) | Controle, n (%) | p‐valor | ||
|---|---|---|---|---|
| 14 | 68 | |||
| c.135G>A | G/G | 13 (92,9) | 59 (86,8) | 0,85 |
| G/A | 1 (7,1) | 9 (13,2) | ||
| c.135G>A | G/G | 14 (100,0) | 68 (100,0) | N/A |
| A/A | 0 | 0 | ||
| c.113_114insA | WT | 13 (92,9) | 57 (83,8) | 0,65 |
| Ins A | 1 (7,1) | 11 (16,2) | ||
| c.74T>A | T/T | 14 (100,0) | 67 (98,5) | 1,00 |
| A/A | 0 (0,0) | 1 (1,5) | ||
| c.141C>A | C/C | 14 (100,0) | 67 (98,5) | 1,00 |
| C/A | 0 (0,0) | 1 (1,5) | ||
| c.435A>C | A/A | 9 (64,3) | 33 (48,5) | 0,44 |
| A/C | 5 (35,7) | 35 (51,5) | ||
| c.435A>C | A/A | 8 (57,1) | 47 (69,1) | 0,58 |
| C/C | 6 (42,9) | 21 (30,9) | ||
| c.613G>A | G/G | 13 (92,9) | 68 (100,0) | 0,38 |
| A/A | 1 (7,1) | 0 (0,0) | ||
| c.770C>T | C/C | 14 (100,0) | 68 (100,0) | N/A |
| C/T | 0 | 0 | ||
| c.936C>T | C/C | 14 (100,0) | 64 (94,1) | 0,80 |
| C/T | 0 (0,0) | 4 (5,9) | ||
| c.1077C>T | C/C | 14 (100,0) | 68 (100,0) | N/A |
| C/T | 0 | 0 | ||
| c.1552C>G | G/G | 14 (100,0) | 65 (95,6) | 0,98 |
| C/C | 0 (0,0) | 3 (4,4) | ||
| c.1552C>G | G/G | 14 (100,0) | 67 (98,5) | 1,00 |
| G/C | 0 (0,0) | 1 (1,5) |
RSCsPN, rinossinusite crônica sem pólipos nasais; n, número de amostras; C, citosina; T, timina; A, adenina; G, guanina; WT, tipo selvagem; N/A, não aplicável.
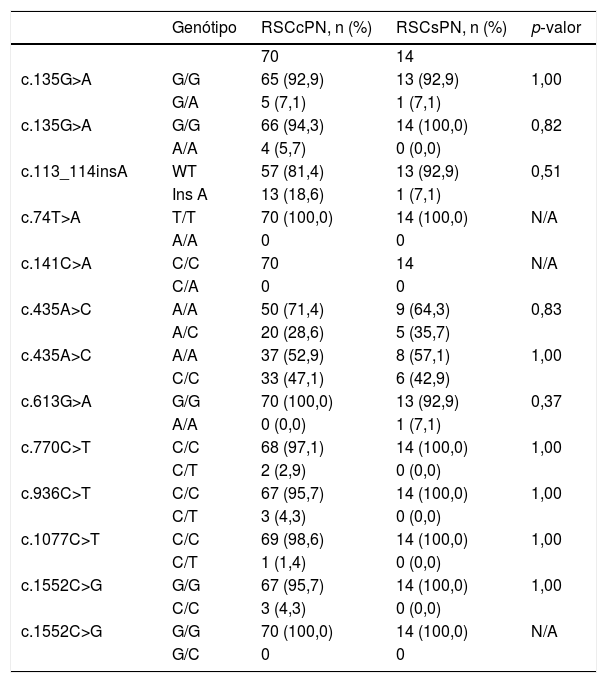
Análise estatística que compara as frequências alélicas em pacientes com rinossinusite crônica com e sem pólipos nasais
| Genótipo | RSCcPN, n (%) | RSCsPN, n (%) | p‐valor | |
|---|---|---|---|---|
| 70 | 14 | |||
| c.135G>A | G/G | 65 (92,9) | 13 (92,9) | 1,00 |
| G/A | 5 (7,1) | 1 (7,1) | ||
| c.135G>A | G/G | 66 (94,3) | 14 (100,0) | 0,82 |
| A/A | 4 (5,7) | 0 (0,0) | ||
| c.113_114insA | WT | 57 (81,4) | 13 (92,9) | 0,51 |
| Ins A | 13 (18,6) | 1 (7,1) | ||
| c.74T>A | T/T | 70 (100,0) | 14 (100,0) | N/A |
| A/A | 0 | 0 | ||
| c.141C>A | C/C | 70 | 14 | N/A |
| C/A | 0 | 0 | ||
| c.435A>C | A/A | 50 (71,4) | 9 (64,3) | 0,83 |
| A/C | 20 (28,6) | 5 (35,7) | ||
| c.435A>C | A/A | 37 (52,9) | 8 (57,1) | 1,00 |
| C/C | 33 (47,1) | 6 (42,9) | ||
| c.613G>A | G/G | 70 (100,0) | 13 (92,9) | 0,37 |
| A/A | 0 (0,0) | 1 (7,1) | ||
| c.770C>T | C/C | 68 (97,1) | 14 (100,0) | 1,00 |
| C/T | 2 (2,9) | 0 (0,0) | ||
| c.936C>T | C/C | 67 (95,7) | 14 (100,0) | 1,00 |
| C/T | 3 (4,3) | 0 (0,0) | ||
| c.1077C>T | C/C | 69 (98,6) | 14 (100,0) | 1,00 |
| C/T | 1 (1,4) | 0 (0,0) | ||
| c.1552C>G | G/G | 67 (95,7) | 14 (100,0) | 1,00 |
| C/C | 3 (4,3) | 0 (0,0) | ||
| c.1552C>G | G/G | 70 (100,0) | 14 (100,0) | N/A |
| G/C | 0 | 0 |
RSCcPN, rinossinusite crônica com pólipos nasais; RSCsPN, rinossinusite crônica sem pólipos nasais; n, número de amostras; C, citosina; T, timina; A, adenina; G, guanina; WT, tipo selvagem; N/A, não aplicável.
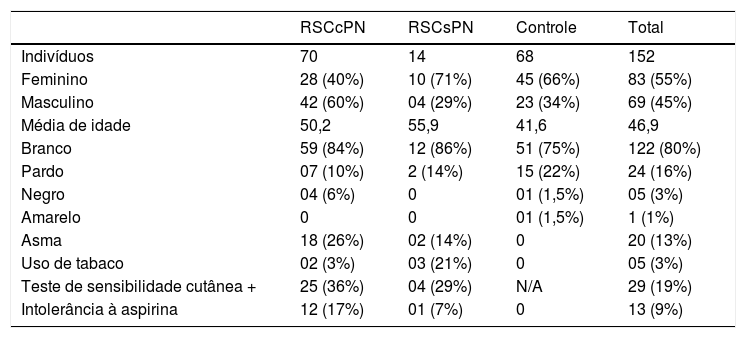
Foram analisadas 152 amostras: 70 de RSCcPN, 14 de RSCsPN e 68 de indivíduos‐controle. As mulheres constituíram 55% dos participantes do estudo e a média de idade foi de 46,9 anos. Quanto à cor da pele, 80% dos indivíduos afirmaram ser brancos, 9% pardos/mulatos, 16% negros, 3% e 1% amarelos. Entre os pacientes com RSC, 13% tinham asma, a maioria deles com PN (90%). O uso de tabaco foi relatado por 3% dos participantes, principalmente no grupo RSCsPN. Dos 19% dos participantes que tiveram testes cutâneos positivos para alergia (teste de puntura ou teste prick), 86% tinham PNs. Dos 9% dos participantes intolerantes à aspirina, 92% eram pacientes com RSCcPN (tabela 4).
Características da população estudada
| RSCcPN | RSCsPN | Controle | Total | |
|---|---|---|---|---|
| Indivíduos | 70 | 14 | 68 | 152 |
| Feminino | 28 (40%) | 10 (71%) | 45 (66%) | 83 (55%) |
| Masculino | 42 (60%) | 04 (29%) | 23 (34%) | 69 (45%) |
| Média de idade | 50,2 | 55,9 | 41,6 | 46,9 |
| Branco | 59 (84%) | 12 (86%) | 51 (75%) | 122 (80%) |
| Pardo | 07 (10%) | 2 (14%) | 15 (22%) | 24 (16%) |
| Negro | 04 (6%) | 0 | 01 (1,5%) | 05 (3%) |
| Amarelo | 0 | 0 | 01 (1,5%) | 1 (1%) |
| Asma | 18 (26%) | 02 (14%) | 0 | 20 (13%) |
| Uso de tabaco | 02 (3%) | 03 (21%) | 0 | 05 (3%) |
| Teste de sensibilidade cutânea + | 25 (36%) | 04 (29%) | N/A | 29 (19%) |
| Intolerância à aspirina | 12 (17%) | 01 (7%) | 0 | 13 (9%) |
RSCcPN, rinossinusite crônica com pólipos nasais; RSCsPN, rinossinusite crônica sem pólipos nasais; N/A, não aplicável.
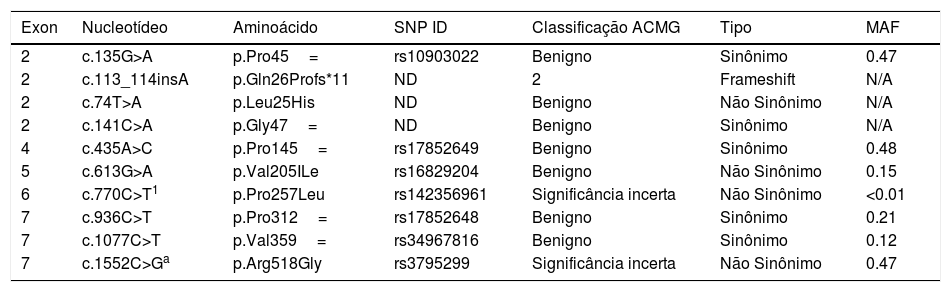
O procedimento de sequenciamento identificou 10 polimorfismos no gene IL22RA1, localizado no cromossomo 1 (tabela 5). Informações sobre a patogenicidade dos polimorfismos c.135G>A, c.74T>A, c.141C>A, c.435A>C, c.770C>T, c.936C>T, e c.1077C>T não estavam disponíveis, mas c.613G>A e c.1552C>G já foram descritos como não patogênicos. A variante c.113_114insA (Gln26Profs*11) é provavelmente patogênica, uma característica típica das mutações frameshift (tabela 6).
Descrição dos polimorfismos encontrados no gene IL22RA1
| Exon | Nucleotídeo | Aminoácido | SNP ID | Classificação ACMG | Tipo | MAF |
|---|---|---|---|---|---|---|
| 2 | c.135G>A | p.Pro45= | rs10903022 | Benigno | Sinônimo | 0.47 |
| 2 | c.113_114insA | p.Gln26Profs*11 | ND | 2 | Frameshift | N/A |
| 2 | c.74T>A | p.Leu25His | ND | Benigno | Não Sinônimo | N/A |
| 2 | c.141C>A | p.Gly47= | ND | Benigno | Sinônimo | N/A |
| 4 | c.435A>C | p.Pro145= | rs17852649 | Benigno | Sinônimo | 0.48 |
| 5 | c.613G>A | p.Val205ILe | rs16829204 | Benigno | Não Sinônimo | 0.15 |
| 6 | c.770C>T1 | p.Pro257Leu | rs142356961 | Significância incerta | Não Sinônimo | <0.01 |
| 7 | c.936C>T | p.Pro312= | rs17852648 | Benigno | Sinônimo | 0.21 |
| 7 | c.1077C>T | p.Val359= | rs34967816 | Benigno | Sinônimo | 0.12 |
| 7 | c.1552C>Ga | p.Arg518Gly | rs3795299 | Significância incerta | Não Sinônimo | 0.47 |
SNP ID, identificação de polimorfismo de nucleotídeo único; MAF, frequência alélica menor; N/A, não aplicável; ND, não descrito.
Combined Annotation Dependent Depletion (CADD) gera pontuações preditivas para variantes de nucleotídeo único em todas as áreas do genoma, inclusive regiões não codificantes.
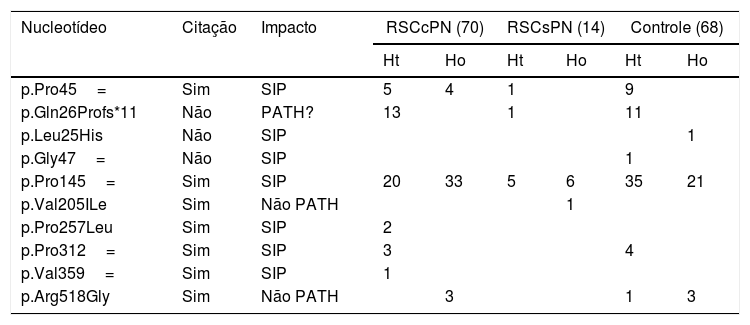
Caracterização dos polimorfismos encontrados no gene IL22RA1 nos diferentes grupos de estudo
| Nucleotídeo | Citação | Impacto | RSCcPN (70) | RSCsPN (14) | Controle (68) | |||
|---|---|---|---|---|---|---|---|---|
| Ht | Ho | Ht | Ho | Ht | Ho | |||
| p.Pro45= | Sim | SIP | 5 | 4 | 1 | 9 | ||
| p.Gln26Profs*11 | Não | PATH? | 13 | 1 | 11 | |||
| p.Leu25His | Não | SIP | 1 | |||||
| p.Gly47= | Não | SIP | 1 | |||||
| p.Pro145= | Sim | SIP | 20 | 33 | 5 | 6 | 35 | 21 |
| p.Val205ILe | Sim | Não PATH | 1 | |||||
| p.Pro257Leu | Sim | SIP | 2 | |||||
| p.Pro312= | Sim | SIP | 3 | 4 | ||||
| p.Val359= | Sim | SIP | 1 | |||||
| p.Arg518Gly | Sim | Não PATH | 3 | 1 | 3 | |||
RSCcPN, rinossinusite crônica com pólipos nasais; RSCsPN, rinossinusite crônica sem pólipos nasais; SIP, sem informação patogênica; Ht, heterozigose; Ho, homozigose; PATH?, provavelmente patogênico; Não PATH, não patogênico.
A variante c.113_114insA (Gln26Profs*11), que não havia sido descrita anteriormente, foi encontrada principalmente na RSCcPN. Essa variação de frameshift pode resultar em uma proteína não funcional, possivelmente contribui para o desenvolvimento da RSC.
Em relação ao exon 4 em heterozigose, apenas a variante c.435A>C (p.Pro145=) apresentou predominância estatisticamente significativa do alelo mutado no grupo controle (51,5%) em comparação ao grupo RSCcPN, sugeriu uma possível proteção contra o fenótipo da RSC. Entretanto, informações sobre a patogenicidade desse polimorfismo não estão disponíveis nos bancos de dados de previsão.
DiscussãoEm uma tentativa de identificar possíveis mecanismos genéticos que desencadeiam a rinossinusite crônica, comparamos os polimorfismos do gene IL22RA1 em amostras de pacientes brasileiros com RSC e em um grupo controle, sem a doença. Identificamos uma nova mutação no gene IL22RA1, do tipo frameshift, c.113_114insA (Q26Pfs*11), que é uma mutação potencialmente patogênica que apareceu principalmente nos grupos de pacientes, mas não houve significância estatística quando comparada ao grupo controle. Além disso, o polimorfismo c.435A>C, em heterozigose, ocorreu com maior frequência no grupo controle do que no grupo RSCcPN, sugeriu que essa variante poderia conferir proteção contra o desenvolvimento de RSC.
Três polimorfismos nunca foram descritos nas bases de dados genéticas analisadas (dbSNP, ExAC): Gln26PProfs*11, p.Leu25His, e p.Gly47=). A variante p.Pro257Leu, detectada em dois pacientes negros com PNs, manteve uma Frequência Alélica Menor (MAF – do inglês Minor Allele Frequency) menor do que 0,01. Como se trata de uma variante caracterizada como de significado incerto e de baixa frequência na população em geral, seria interessante fazer ensaios funcionais para confirmar sua patogenicidade.
O estudo conduzido por Ramanathan et al.13 foi o primeiro a demonstrar diminuição da expressão do gene IL22RA1 em indivíduos com RSCcPN em comparação com indivíduos sem pólipos e grupo controle, mas nenhuma explicação foi proposta para essa diferença. Um estudo com pacientes canadenses propôs polimorfismos do gene IL22RA1 como uma causa potencial para RSC.9 No entanto, os polimorfismos de IL22RA1 aqui descritos são diferentes daqueles descritos por Mfuna‐Endam et al. (2009).5 Isso pode ser devido às diferentes populações dos quais foram amostrados, aos diferentes fatores ambientais aos quais as populações foram expostas ou ainda ao menor número de pacientes analisados em nosso estudo.
Não investigamos como as mutações observadas aqui poderiam contribuir para o desenvolvimento da RSC, com ou sem PNs. Sabe‐se que pacientes com RSC apresentam menor expressão de IL‐22, potencialmente causa disfunção da barreira epitelial e alterações na resposta de Th1, e o desequilíbrio inflamatório entre Th1 e Th2 pode contribuir para o desenvolvimento da RSC. Variações no gene IL22RA1 podem desregular os mecanismos de imunidade inata, levar à diminuição de mediadores inflamatórios específicos. O comprometimento do complexo heterodímero que inclui IL‐10R2, IL‐20 e IL‐24 é outro mecanismo que pode dificultar a transdução de sinal e a ativação da transcrição (via JAK/STAT), levar à desregulação dos níveis de citocinas pró‐inflamatórias.
Apesar de resultar em alterações que podem levar a mudanças na configuração das proteínas, a incidência dos polimorfismos detectados foi baixa. Estudos adicionais são necessários, em coortes maiores, para diminuir a probabilidade de que esses achados sejam aleatórios, garantir sua reprodução e definir o impacto desses polimorfismos na doença em questão.
ConclusõesNeste estudo, identificamos um novo polimorfismo associado positivamente à RSCcPN, a variante c.113_114insA (Q26Pfs*11). Além disso, um polimorfismo no exon 4 foi significantemente mais frequente no grupo controle do que no grupo RSCcPN (c.435A>C em heterozigose), sugeriu uma proteção potencial contra o fenótipo da RSC.
Conflitos de interesseOs autores declaram não haver conflitos de interesse.
A todos os funcionários do Departamento de Genética e do Centro de Genômica Médica do Hospital das Clínicas da Faculdade de Medicina de Ribeirão Preto da Universidade de São Paulo, Brasil, que de alguma forma contribuíram para esta pesquisa.
Como citar este artigo: Dinarte VR, Silva WA, Baccarin ARD, Tamashiro E, Valera FC, Anselmo‐Lima WT. Association of interleukin 22 receptor subunit alpha 1 gene polymorphisms with chronic rhinosinusitis. Braz J Otorhinolaryngol. 2021;87:505–11.
A revisão por pares é da responsabilidade da Associação Brasileira de Otorrinolaringologia e Cirurgia Cérvico‐Facial.